2022年数理科学学院第七期研究生论坛如期举行。5月5日上午9:00-12:00,RCNS全体成员和各位导师共同参加。在这次组会上,首先由1名研一学生汇报自己的研究进展,然后由2名研三的学生汇报自己的毕业论文,最后老师与同学们对汇报内容进行学术探讨,并对存在的问题给出相应的指导和建议。
翟立凯:本文研究了具有不同时间尺度的随机FitzHugh–Nagumo系统的平均原理。在适当的条件下,证明了该耦合系统消除快速变量的平均方程的存在性,从而将该系统简化为一个具有修正系数的单一随机普通方程。此外,慢分量对平均方程解的收敛速度为1/2阶。

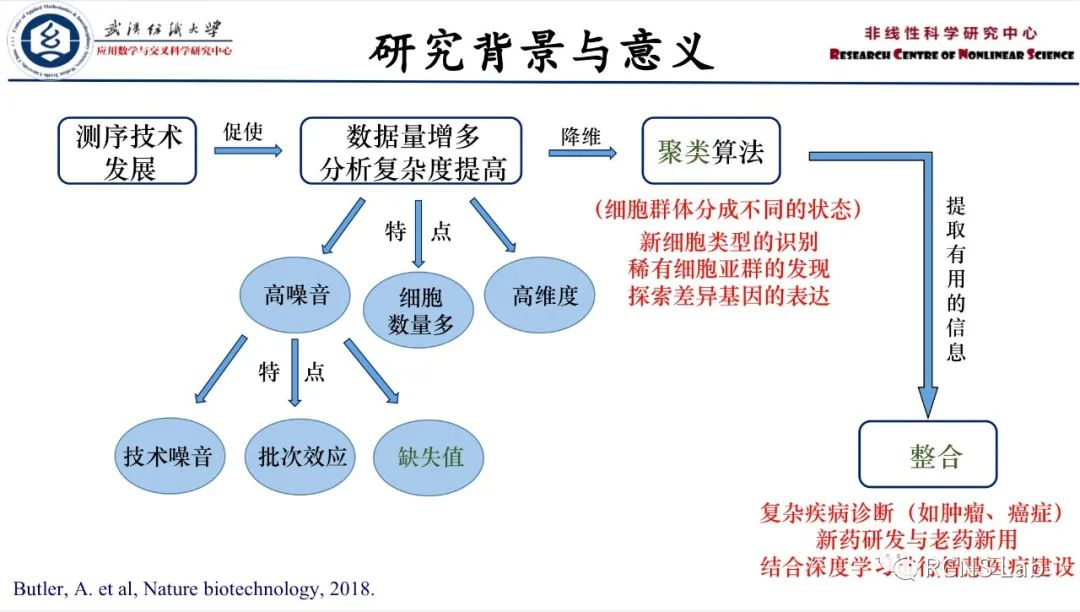
曹文洁:高通量的单细胞转录组测序技术能够以单细胞分辨率表征转录图谱,更好地揭示未知生物的多样性。然而,由于RNA转录组的数量少、基因表达模式的随机性、细胞的低捕获和低测序效率产生大量的技术和生物噪音,导致scRNA-seq具有高维度、稀疏性、具有大量缺失值以及受批次效应影响的特点。正是scRNA-seq数据中存在的大量缺失值,掩盖了基因间的关系,阻碍了下游分析。因此,对于scRNA-seq数据缺失值的插补,细胞类型和巨大细胞数量的精确快速分类仍是一项巨大挑战。基于生物数据,对其缺失值插补与分类进行研究,主要工作包含如下几方面:1、对单细胞数据缺失值插补方法进行综述研究。通过算法原理与优缺点的比较和分析,为针对特定问题与数据在插补方法的选择上提供建议,对数据的下游功能分析具有基础性研究意义。然后,针对数据稀疏、存在大量缺失值的问题,提出了一种基于多个编码解码器和注意力网络的深度神经网络模型(AMEDNN),通过实验验证了该模型的有效性。2、基于生物序列数据分类算法的研究。针对RNN存在的记忆能力不足以及梯度反向传播困难等问题提出一种新的算法SS-RNN。3、针对scRNA-seq数据分类提出了六种基于集成学习的分类模型,EL-KNN、EL-LDA、EL-SVM、EL-NB、EL-DT和EL-HW。4、基于图注意力网络的单细胞转录组测序数据缺失值插补与聚类算法scGATv2。该框架通过三种不同的自动编码器的迭代,来挖掘细胞间的潜在关系。本工作主要创新之处在于融入图注意力变分自动编码器,不仅能保持原本的拓扑结构,还可以通过注意力机制自动的学习和优化细胞间的连接关系。
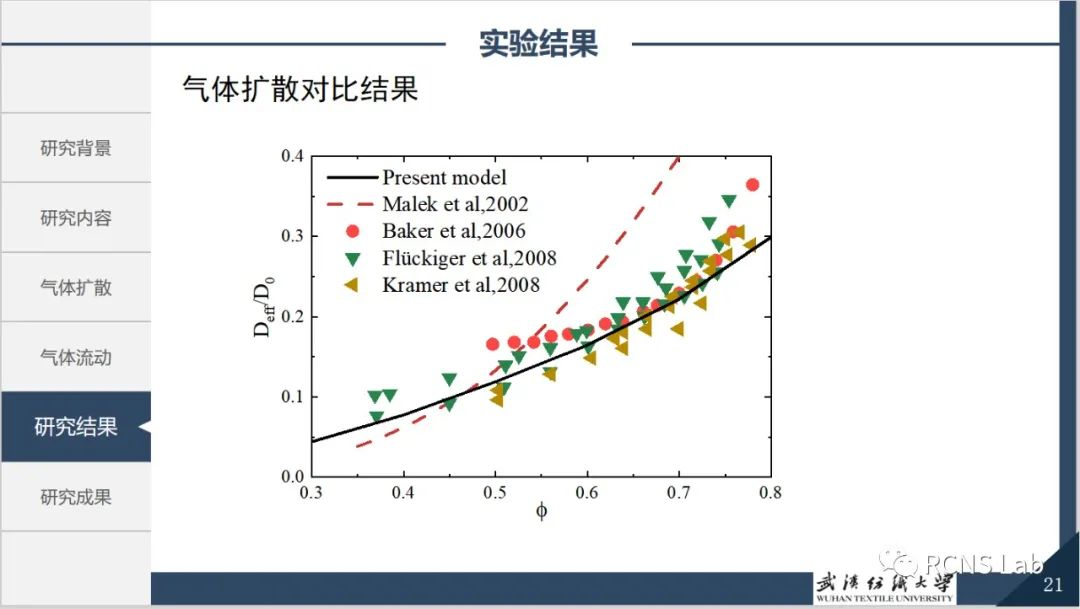
尹作壮:由于多孔介质内部孔隙结构的复杂性和随机性,探索气体输运过程时往往忽略了内部孔隙或通道的表面形貌。因此拟基于分形几何理论与蒙特卡罗方法,重点模拟具有粗糙形貌的多孔介质中气体输运的详细过程,主要包括气体扩散和气体流动,分析多孔介质结构参数,特别是孔隙表面粗糙度对气体输运特性的详细影响机理。
— 学生汇报照片展示 —
