应用数学与交叉科学研究中心生物信息学团队于2023年12月12日举行第三十五期研究生论坛,小组全体成员和各位导师共同参加。在这次论坛上,由两名研一学生、一名研二学生和一名研三学生分别汇报自己的研究进展,然后老师与同学们对汇报内容进行学术探讨,并对存在的问题给出相应的指导和建议。
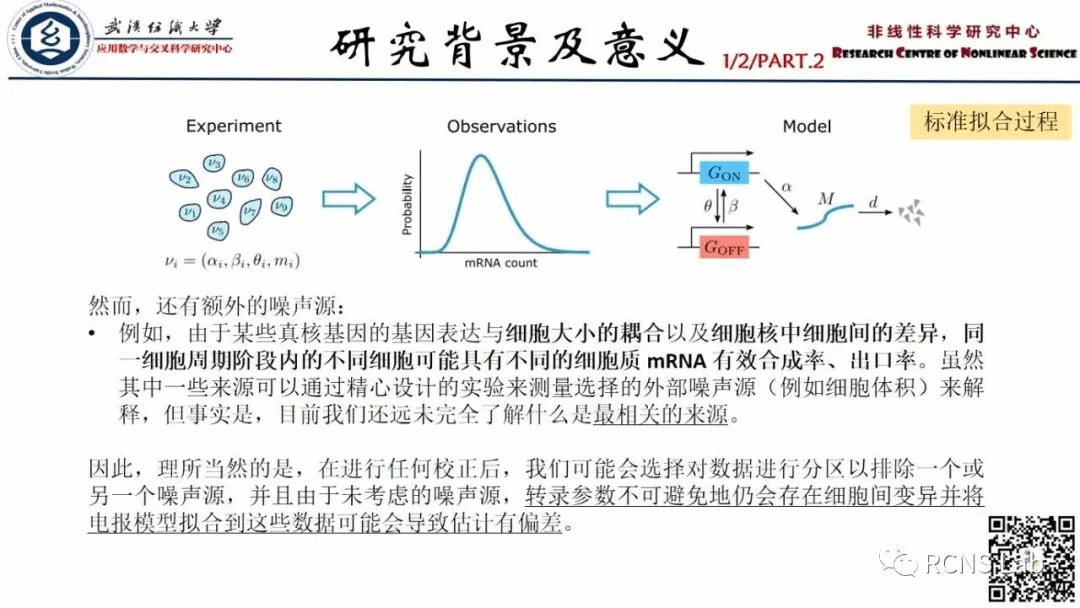
张秋玉:本次组会汇报了一篇文献《Quantifying and correcting bias in transcriptional parameter inference from single-cell data》。每个细胞mRNA计数的快照分布可以使用单分子荧光原位杂交或单细胞RNA测序来测量。这些分布通常适合于两态电报模型的稳态分布,以估计感兴趣的基因的三个转录参数:mRNA合成率,开启率(开启状态为活性转录状态)和关闭率。该模型假设没有外部噪声,即参数在细胞之间不变化,因此估计的参数应被理解为近似于总体中的平均值。这种近似的准确性目前尚不清楚。在这里,作者发展了一个解释了使用标准电报模型从单细胞数据推断参数时估计偏差的大小和符号的理论。发现特定的偏倚特征取决于外部噪声的来源(该参数在细胞间变化最大)和转录活性的模式。因此,在输入到基于电报模型的推理框架之前,对数据进行外部噪声校正是很重要的,因为这种类型的噪声可能会显著影响参数估计。
韩长锋:本次汇报了关于《基于轨迹推断的细胞命运决策机理研究》的写作框架。特别地,对从数据出发经过轨迹推断过程后如何再进一步的研究细胞命运决策的问题,与各位老师以及同学进行了深入的讨论和交流。
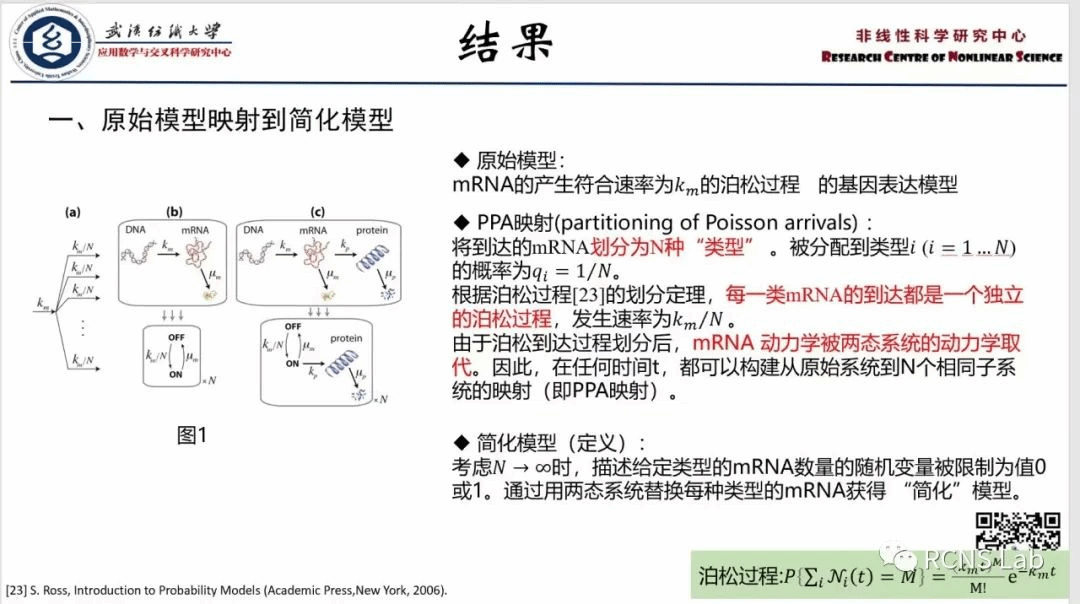
郭贞贞:本次汇报了一篇文献《Exact protein distributions for stochastic models of gene expression using partitioning of Poisson processes》。基因表达的随机性导致基因相同的细胞群体中蛋白质水平的波动。这种波动可导致克隆群体的表型变异,因此,人们对使用随机模型量化基因表达中的噪声有相当大的兴趣。然而,除了最简单的模型外,获得蛋白质分布的精确分析结果是个复杂的工作。在这里,文章调用泊松过程的划分特性来构建一个映射,简化了基因表达随机模型的分析。通过使用基于启动子调控的模型中mRNA分布的结果,该映射导致精确的蛋白质分布。文章中使用这种方法导出了基因表达的基本两阶段模型稳态的时间依赖分布的精确分析结果。此外,文章还展示了扩展两阶段基本模型之后的精确分布,包括转录后和翻译后调控的影响。
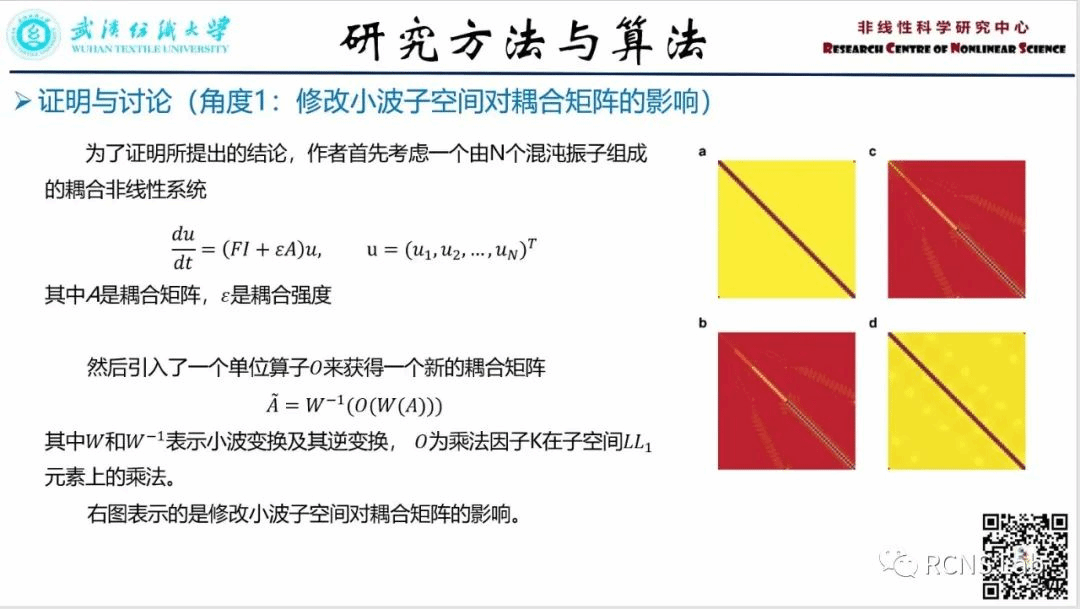
张春焕:本次汇报了一篇文献《Tailoring Wavelets for Chaos Control》。小波理论作为一种分析工具,在非线性动力学中得到了广泛的应用,但其使用仅限于分析和表征。直接使用小波作为控制系统动力学的基础还没有得到开发,本文的目的就是介绍一个使用小波进行混沌控制和同步的新范式,研究发现,通过修改耦合矩阵小波子空间的一小部分就可以显著增强混沌系统中同步流行的横向稳定性。
— 学生汇报照片展示 —



