近期,数理科学学院大数据分析与处理科研团队成员江健教授与其合作者在药物设计及开发方向取得重要研究进展,相关成果以“TIDAL: Topology-Inferred Drug Addiction Learning”为题在该领域国际知名期刊Journal of chemical information and modeling上发表。该论文的第一作者为团队硕士研究生朱在亮,江健教授为第一通讯作者。
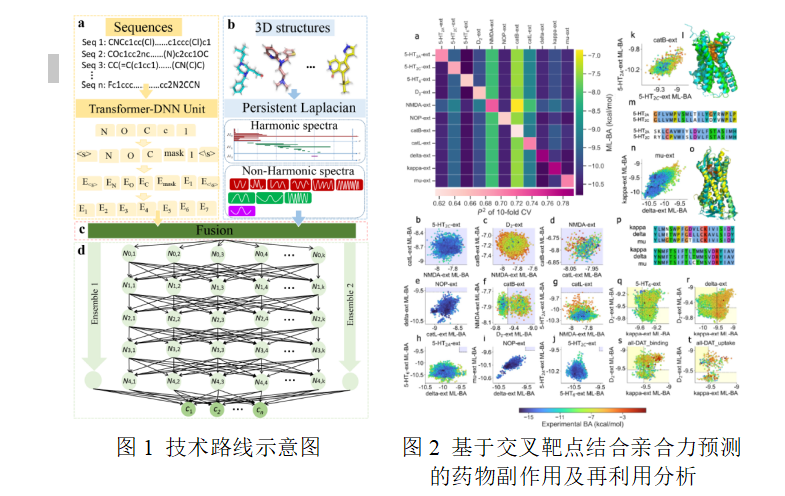
该论文主要利用拓扑学和图论,结合深度学习算法研究药物致瘾问题。药物致瘾是一个全球社会和健康危机问题,抗上瘾药物的研发仍然面临巨大挑战。该论文利用拓扑学中的霍奇理论,发展新的生物分子的数学表征方法,结合深度学习算法对药物上瘾相关分子数据集进行建模和分析。通过预测38个数据集中上瘾候选分子的交叉靶点结合亲和力,分析它们的副作用和潜在的再利用功能,发现基于霍奇理论的分子表征方法相比其他经典分子指纹和基于机器学习的学习表征方法具有更高的预测准确度,对抗上瘾药物的开发具有实际指导意义。
另悉,该团队谭雅岚博士与其合作者在RNA三维结构预测方面取得新突破。相关成果以“cgRNASP: coarse-grained statistical potentials with residue separation for RNA structure evaluation”为题在该领域国际知名期刊NAR的姊妹刊——NAR Genomics and Bioinformatics上发表。该论文的第一作者为团队谭雅岚博士,张本龚教授为共同通讯作者。

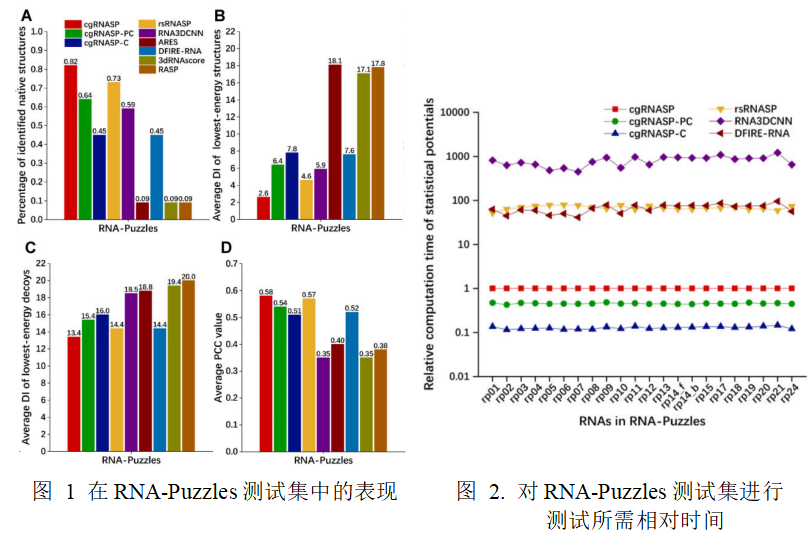
对RNA理论模型预测得到的候选结构进行高性能评估是实现 RNA 三维结构高精度预测中至关重要的步骤。该论文在全原子评估统计势(rsRNASP; Tan et al,Biophysical Journal,2022)的基础上,通过选取 C、P、N 等原子来代表核苷酸中重要的基团,并进一步细致考虑RNA链上近邻和次近邻碱基间的相互作用提出了粗粒化统计势(cgRNASP、cgRNASP-PC和 cgRNASP-C)。针对来自于不同模型预测的结构集,cgRNASP的性能与rsRNASP相当;而其效率比现有全原子统计势(rsRNASP、RNA3DCNN以及 DFIRE-RNA)高几十倍。cgRNASP的提出为评估RNA预测结构提供了高效高性能的新工具。cgRNASP 以 及 相 关 数 据 库 已 开 源 供 同 行 下 载 使 用[https://github.com/Tan-group/cgRNASP]。
应用数学与交叉科学研究中心大数据分析与处理科研团队长期从事单细胞测序数据分析、蛋白质分子结构预测、药物虚拟筛选及深度学习图像处理研究。近年来,该团队承担国家自科基金项目6项,其中面上2项,省部级项目5项,发表高水平SCI论文60余篇。
论文链接:https://doi.org/10.1021/acs.jcim.3c00046
https://doi.org/10.1093/nargab/lqad016