RCNS实验室在3月31日上午9:00-12:00举行了2022年数理科学学院第二期研究生论坛,RCNS全体成员和各位导师共同参加。在这次组会上,首先由2名研一学生和2名研二学生汇报自己的研究进展,然后老师与同学们对汇报内容进行学术探讨,并对存在的问题给出相应的指导和建议
木子淳(线上汇报):本次汇报了一篇名为《Martini Coarse-Grained Force Field: Extension to DNA》的Martini 粗粒化DNA模型。该模型系统地参数化了一个可以与Martini力场兼容的DNA粗粒度(CG)模型。该模型将每个核苷酸映射为6到7个CG珠,并采取Martini力场的标准势能函数(包括成键势和非成键势),通过计算PMF来获得相应的碱基堆积势以及碱基配对的氢键势。以上的相互作用势仅适合单链DNA (ssDNA)的原子模拟,对于双链DNA (dsDNA)采取弹性网络用于保留dsDNA和其他特定的DNA构象。本文提出的Martini DNA模型,展现了单个碱基、ssDNA和dsDNA以及DNA -蛋白质复合物的性质。该模型开创了模拟DNA与其他生物大分子相互作用的方法。
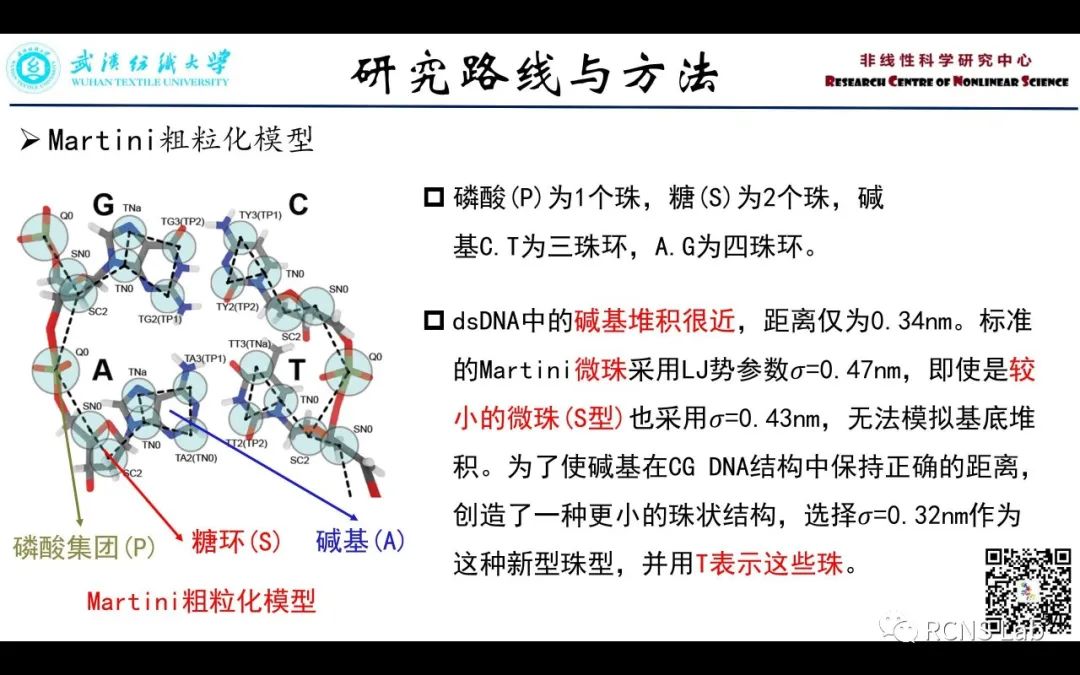
熊珍珍(线上汇报):该文献是考虑基因和细胞之间的关联提出的基于协同矩阵分解的单细胞测序数据插补方法。该方法首先将所使用的数据集按照细胞标记的可信度分为金银铜标准,接下来通过三个相似度度量:欧几里得距离,切比雪夫距离和皮尔逊相关系数,分别计算细胞相似性矩阵和基因相似性矩阵。CMF-Impute算法的目的,就是找出细胞特征矩阵和基因特征矩阵,使两个特征矩阵的乘积最接近原始矩阵,以此重新定义基因表达矩阵,最终达到数据插补目的。
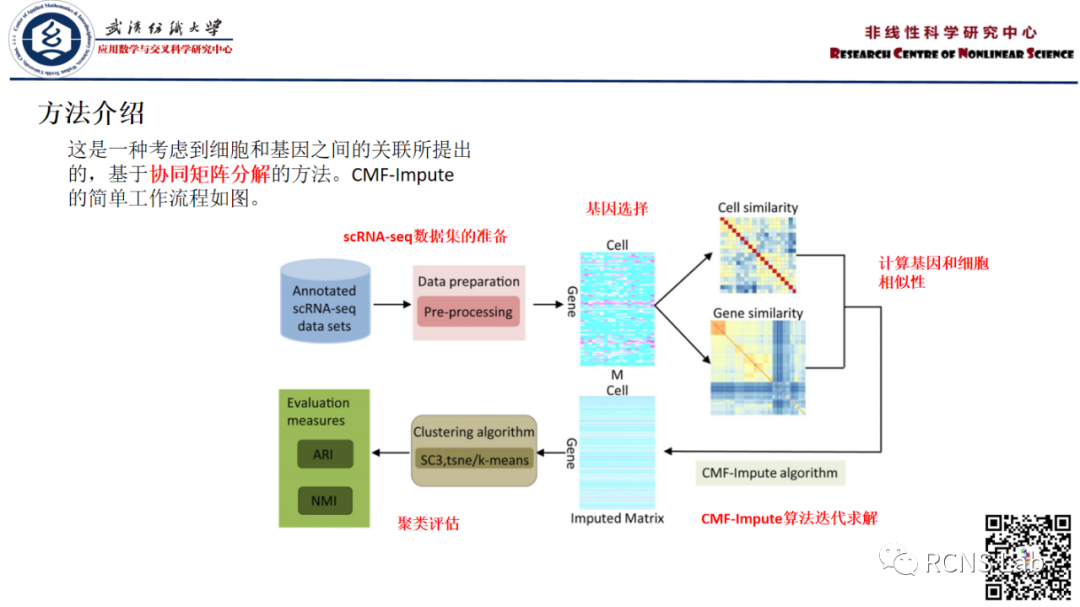
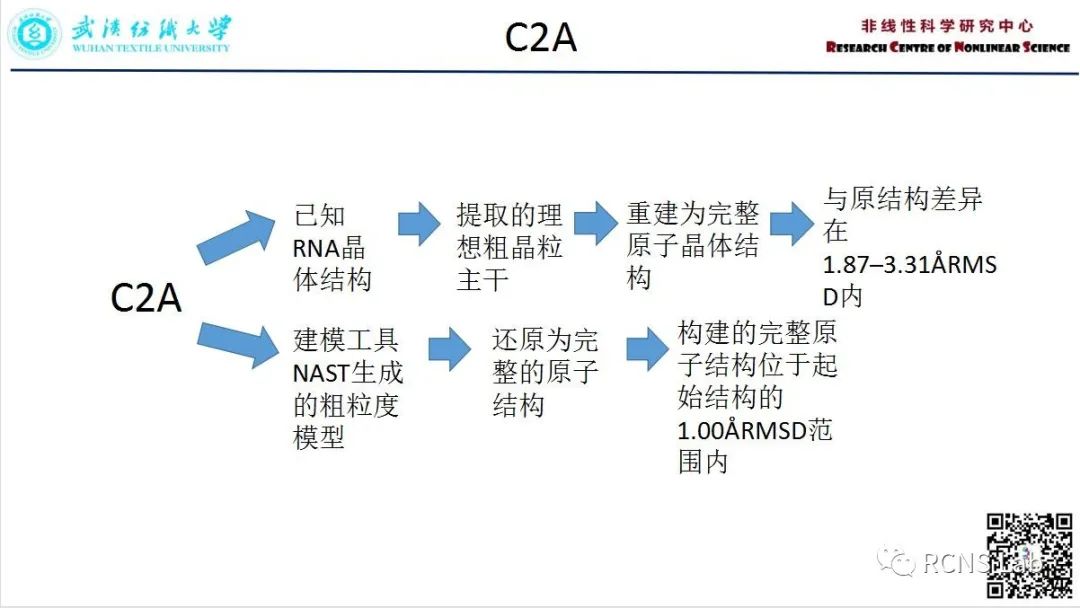
吴昊:本次汇报的文献为《C2A:基于已知的全原子还原粗粒化RNA3D结构》。使用粗粒度方法对RNA 3D结构进行建模的方法不断发展,需要在低分辨率和高分辨率建模方法之间架起桥梁。虽然粗粒度模型包含拓扑信息,但缺乏原子细节,这限制了它们在某些应用中的实用性。C2A可以使用任何基于原子的粗粒度结构模板作为输入,并提供一个全原子、能量最小化的结构作为输出。利用RNA晶体结构的几何信息来重建完整的原子细节,已经在70到244个残基大小的分子的理想粗颗粒结构上,以及在NAST生成的每残基一点粗颗粒tRNA模型上测试了C2A方法。除了提高粗粒度建模方法的实用性外,该工具还有潜力将计算成本高但精度高的全原子建模方法与快速但细节差的粗粒度建模方法联系起来。
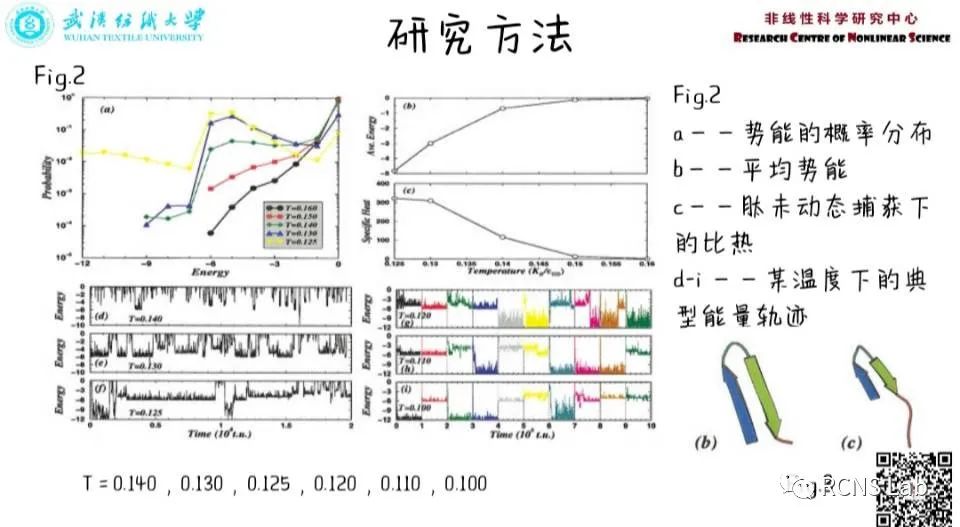
袁洁:在本次汇报中分享了一篇名为《Mechanism for the α-Helix to β-Hairpin Transition》的文献,本文对α-Helex和β-Hairpin的转换进行解析。富含α-Helix的蛋白质聚集成富含β-Hairpin的淀粉样纤维,与一些致命性疾病有关,本文主要对聚丙氨酸进行离散分子动力学模拟,根据在温度、环境的改变下带来的蛋白质结构状态改变。采用Four-Bead Model简化模型,从多角度分析β-发夹中间体的存在性,以及结构状态改变的条件。
— 学生汇报照片展示 —



