RCNS实验室在3月24日上午9:00-12:00举办了第一期研究生论坛,RCNS全体成员和各位导师共同参加。在这次组会上,首先由3名研一学生和1名研二学生汇报自己的研究进展,然后老师与同学们对汇报内容进行学术探讨,并对存在的问题给出相应的指导和建议。
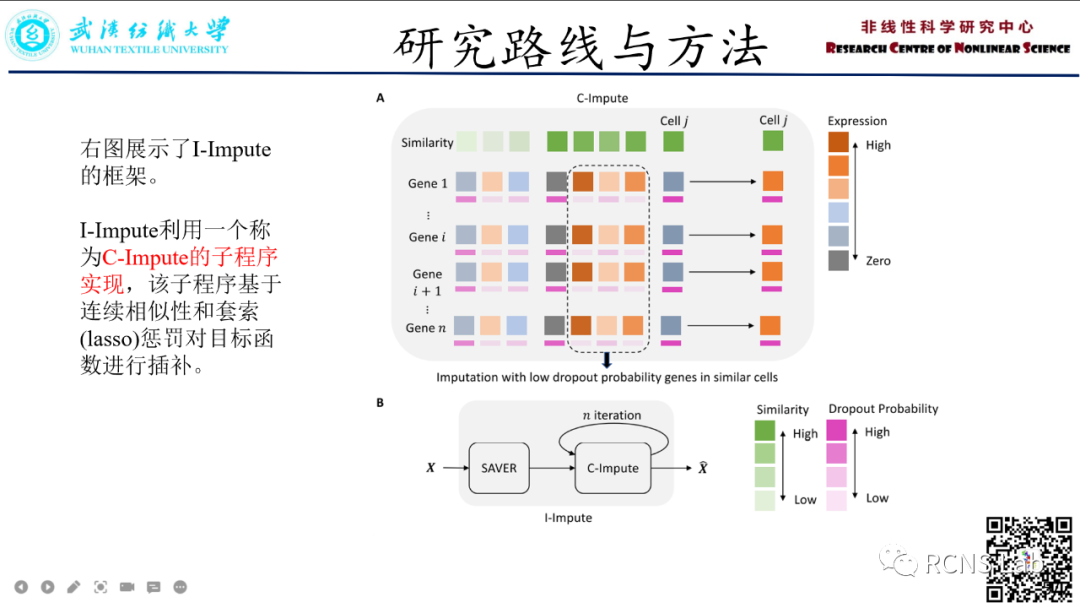
韩长锋:在本次汇报中分享了一篇名为《I-Impute:a self-consistent method to impute single cell RNA sequencing data》的文章,本文对插补一致性提出了一种新的解释。作为一个可靠的插补工具,应该假设其输出不包含丢失或错误。希望插补工具的输出是一致的:如果我们删除一些条目之后再次将插补后的输出提供给插补工具,那么该工具应该能够再现这些条目。我们把这种特性称为自洽(self-consistency)。
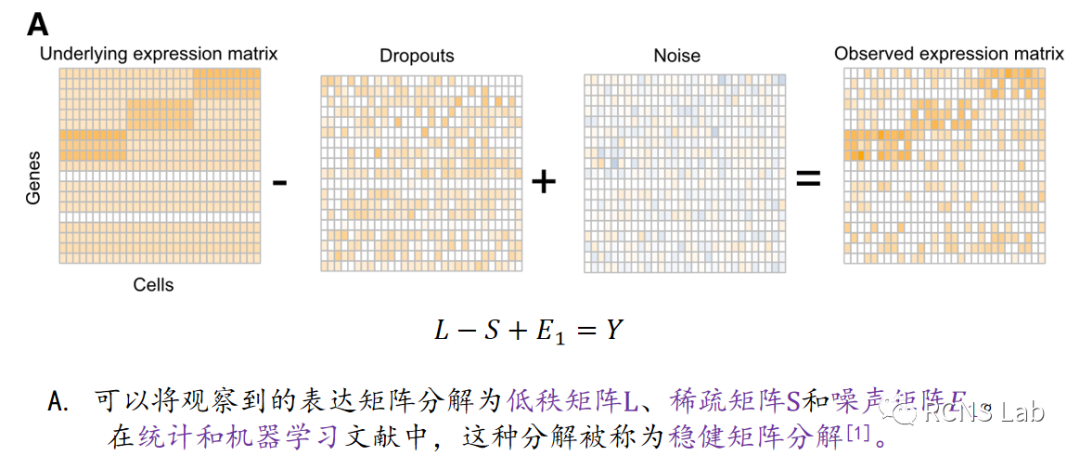
王梦园:本次汇报的文献为《scRMD:基于稳健矩阵分解的单细胞RNA-seq数据插补》,单细胞RNA测序(scRNA-seq)技术能够在单细胞分辨率下对整个转录组进行分析,然而,scRNA-seq常常不能捕捉到表达的基因,导致突出的“缺失”现象。这些缺失在下游分析中造成了许多问题。本文提出的方法scRMD可以将观察到的表达矩阵分解为低秩矩阵L、稀疏矩阵S和噪声矩阵E1,通过使用ADMM(交替方向乘子法)求解最小化噪声项这一优化问题来估计L和S,从而实现单细胞转录组测序数据中的缺失值插补。实验结果表明,即使是在缺失率超过百分之八十的实际数据中,scRMD执行的效果也相当好。
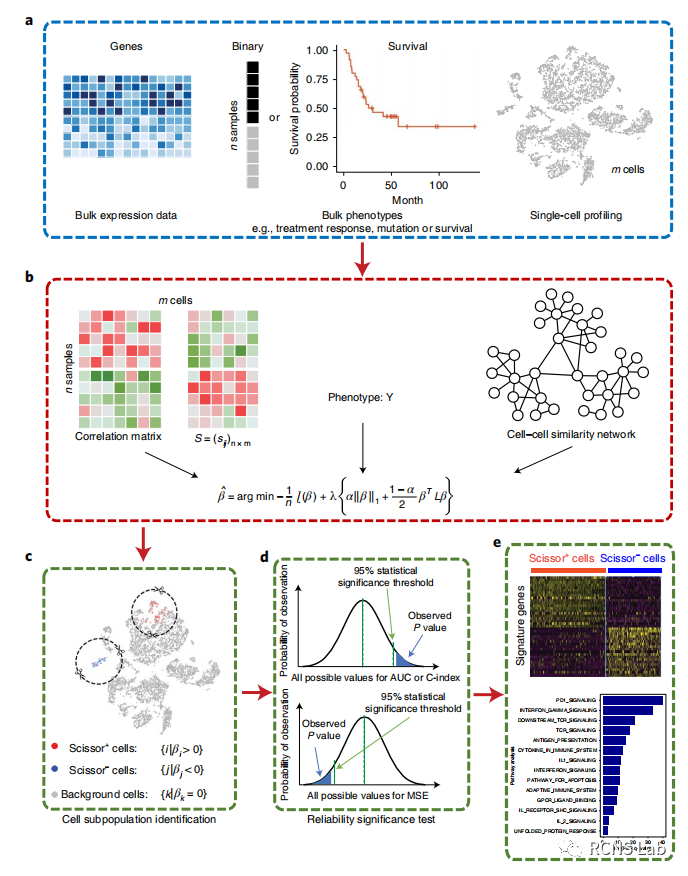
李成:本次汇报的文献为《Identifying phenotype-associated subpopulations by integrating bulk and single-cell sequencing data》单细胞RNA测序(scRNA-seq)可以区分异质组织中的细胞类型、状态和谱系。然而,当前的单细胞数据不能直接将细胞团与特定的表型联系起来。这里我们介绍Scissor,这是一种从与给定表型相关的单细胞数据中识别细胞亚群的方法。Scissor通过首先量化每个单个细胞和每个批量样本之间的相似性,整合了表型相关的批量表达数据和单细胞数据,然后在样本表型的相关矩阵上优化回归模型,以识别相关的亚群。应用于肺癌scRNA-seq数据集,Scissor确定了与较差存活率和TP53突变相关的细胞亚群。在黑色素瘤中,Scissor发现了与免疫治疗反应相关的低PDCD1/CTLA4和高Tcf7表达的T细胞亚群。除了癌症,Scissor在解释面肩肱骨肌营养不良症和阿尔茨海默病数据集方面也是有效的, Scissor通过利用表型和批量组学数据集,从单细胞分析中识别生物学和临床相关的细胞亚群。
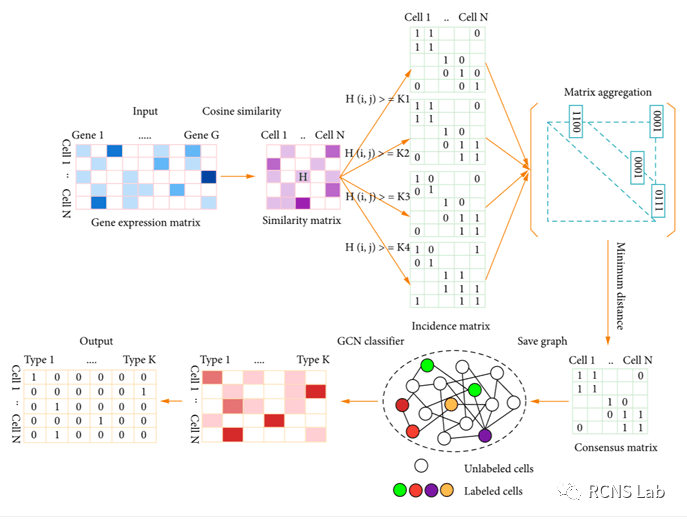
王婷:本次汇报的文献为《SCMAG: A Semisupervised Single Cell Clustering Method Based on Matrix Aggregation Graph Convolutional Neural Network》,聚类分析是单细胞数据挖掘最重要的技术之一。广泛应用于不同基因序列的划分、功能基因的鉴定、新细胞类型的检测等。传统的无监督聚类方法虽然不需要标签数据,但原始数据的分布、超参数的设置等因素都会影响聚类算法的有效性。虽然在某些情况下某些细胞的类型是已知的,但如果充分利用有关这些细胞的先验信息,则有望实现高精度。而SCMAG(一种基于矩阵聚合图卷积神经网络的半监督单细胞聚类方法),它充分考虑了单细胞数据的先验信息,有效提升了聚类精度。SCMAG的工作流程如下所示,输入是一个基因表达矩阵;该算法包括四个步骤:(1)通过余弦相似度公式计算相似度矩阵;(2)通过阈值判断关联矩阵;(3)通过矩阵聚合方法构建共识矩阵;(4)将共识矩阵保存为图;(5)最后,将图作为GCN分类器的输入进行训练。